映射新的数据点
在前面的两个例子中,我们将原始的数据集映射到新的特征空间。不过在实际应用中,我们常常需要将多个数据集转换,比如训练集和测试集,还有可能在训练好模型后,又收集到的新数据。在本节,你将学习如何将不属于训练集的数据进行映射。
还记得在标准PCA中,我们通过计算 转换矩阵*输入样本,得到映射后的数据。转换矩阵的每一列是我们从协方差矩阵中得到的k个特征向量。现在,如何将这种思路应用到核PCA?在核PCA中,我们得到的特征向量来自归一化的核矩阵(centered kernel matrix),而不是协方差矩阵,这意味着样本已经被映射到主成分轴.因此,如果我们要把一个新样本
映射到主成分轴,我们要按照下式:
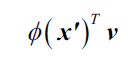
上式怎么算?当然不好算,好在我们还有核技巧,所以可以避免直接计算。
和标准PCA不同的是,核PCA是一种基于内存的方法,这是什么意思呢?意思是每次对新样本进行映射时就要用到所有的训练集。因为要计算训练集中每个样本和新样本之间的RBF核(相似度):
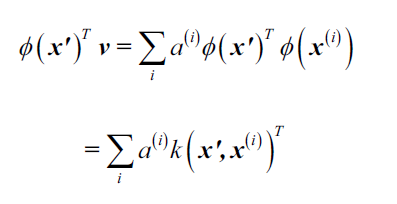
其中,核矩阵的特征向量
和特征值
满足条件:
。
计算每一个训练集样本和新样本的后,我们必须用特征值对特征向量做归一化。所以呢,我们要修改一下前面实现的RBF PCA,能够返回核矩阵的特征向量:
from scipy.spatial.distance import pdist, squareform
from scipy import exp
from scipy.linalg import eigh
import numpy as np
def rbf_kernel_pca(X, gamma, n_components):
"""
RBF kernel PCA implementation.
"""
# Calculate pairwise squared Eculidean distances
sq_dists = pdist(X, 'sqeuclidean')
mat_sq_dists = squareform(sq_dists)
#Compute the symmetric kernel matrix
K = exp(-gamma * mat_sq_dists)
#Center the kernle matrix
N = K.shape[0]
one_n = np.ones((N, N)) / N
K = K - one_n.dot(K) - K.dot(one_n) + one_n.dot(K).dot(one_n)
#Obtaining eigenpairs from the centered kernel matrix
eigvals, eigvecs = eigh(K)
alphas = np.column_stack((eigvecs[:, -i]
for i in range(1,n_components+1)))
#Collect the corresponding eigenvalues
lambdas = [eigvals[-i] for i in range(1, n_components+1)]
return alphas, lambdas
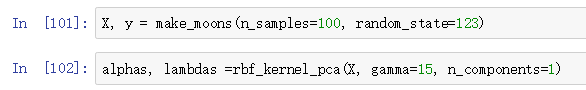
现在,我们创建一个新的半月形数据集,然后用更新过的核PCA将其映射到一维子空间:
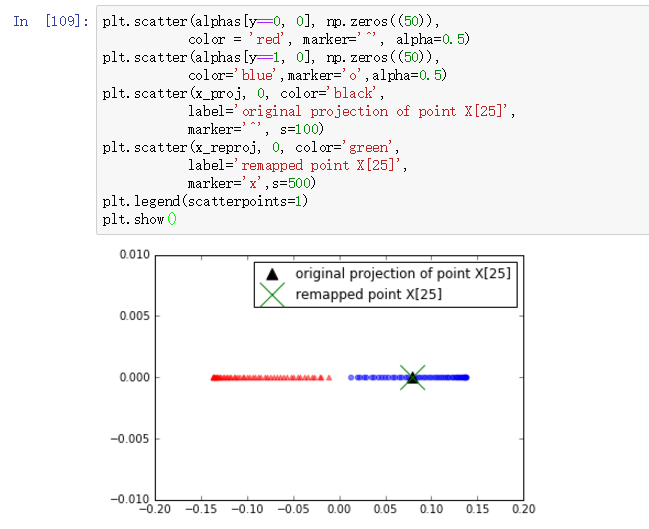
为了检验对于新数据点的映射表现,我们假设第26个点时新数据点,我们的目标就是将这个新数据点进行映射:
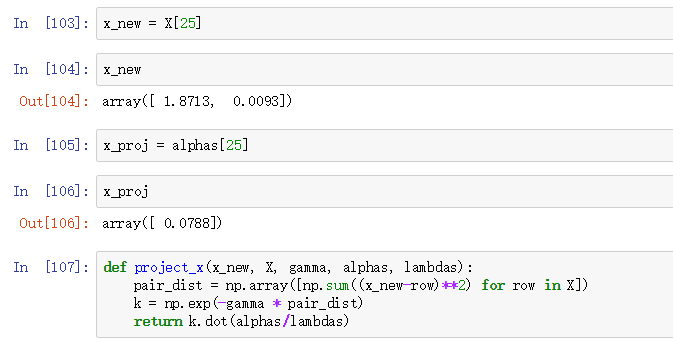
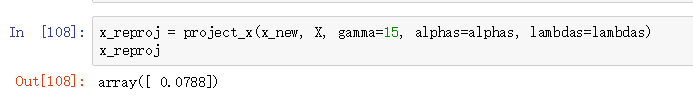
使用project_x函数,我们能够对新数据样本进行映射:
最后,我们将第一主成分进行可视化: